今までデータをあれこれいじったり記述統計出したりするのはExcelで色々やってからRにいれて分析という流れでした。dplyr使ったら便利そう…というのはなんとなくわかっていたんですが,自分がよく扱うようなデータセットの構造に対してどうやって使えば今までやってたことが楽になるのかいまいちイメージがわかずにずっと放置していました。最近色々あってようやく基礎中の基礎だけを覚えたので以下メモ書きです。
まずはサンプルのデータを作ります
#事前・事後・遅延事後でCAFのデータを取ってるというデザイン
pre.fluency <-rnorm(50,mean=10,sd=2)
pre.accuracy<-rnorm(50,mean=7,sd=2)
pre.complexity<-rnorm(50,mean=4,sd=2)
post.fluency<-rnorm(50,mean=14,sd=4)
post.accuracy<-rnorm(50,mean=10,sd=3)
post.complexity<-rnorm(50,mean=7,sd=2)
delayed.fluency<-rnorm(50,mean=12,sd=2)
delayed.accuracy<-rnorm(50,mean=8,sd=1)
delayed.complexity<-rnorm(50,mean=6,sd=1)
#それぞれの列を横にくっつける
dat<-cbind(pre.fluency,pre.accuracy,pre.complexity,post.fluency,post.accuracy,post.complexity,delayed.fluency,delayed.accuracy,delayed.complexity)
dat<-as.data.frame(dat) #データフレーム型に変換
dat$subject<-rep(1:50) #実験参与者のID列をつける
とりあえずこんな感じでいまデータを持ってるとする
head(dat)
## pre.fluency pre.accuracy pre.complexity post.fluency post.accuracy
## 1 11.916630 7.480500 4.644798 8.431656 13.070707
## 2 10.719751 7.090714 6.566487 13.652658 11.462576
## 3 11.980852 6.182161 5.731289 16.580042 11.287683
## 4 11.109400 7.256950 5.897070 16.966183 9.497555
## 5 10.488391 5.449349 2.673285 15.778588 9.994575
## 6 9.076144 8.621538 2.122296 9.465845 7.577611
## post.complexity delayed.fluency delayed.accuracy delayed.complexity
## 1 8.639772 12.961946 7.740613 7.042510
## 2 6.397390 9.921947 7.520258 6.951231
## 3 6.917590 10.478401 9.378301 4.972710
## 4 5.897965 6.738074 8.983224 5.045168
## 5 6.214526 10.694636 8.510670 5.184044
## 6 7.644466 8.666421 8.863990 4.719461
## subject
## 1 1
## 2 2
## 3 3
## 4 4
## 5 5
## 6 6
まずは縦横変換
参考までに以前tidyrの紹介を書いたブログ→変数が多い研究デザイン系の縦横変換(tidyr)
library(tidyr)
dat%>%
gather(key=variable,value=score,-subject)%>%
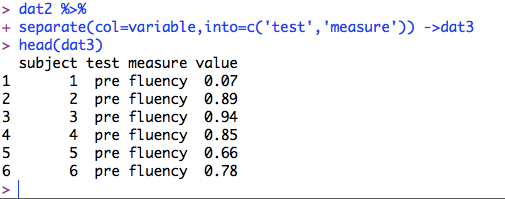
separate(col=variable,into=c('test','measure'))->dat2
head(dat2) #test, measure, scoreという列ができてるか確認
## subject test measure score
## 1 1 pre fluency 11.916630
## 2 2 pre fluency 10.719751
## 3 3 pre fluency 11.980852
## 4 4 pre fluency 11.109400
## 5 5 pre fluency 10.488391
## 6 6 pre fluency 9.076144
悩み
でもさーこうやってlong型にしちゃうとさー例えば事前のときの流暢さの指標の平均値とか出せないじゃん?wide型になってたら全部横にならんでるから列ごとに平均求めればいいだけじゃん?
long型になってても層ごとの記述統計出せちゃう
そう,dplyrならね
library(dplyr)
dat2%>%
group_by(test)%>%
group_by(measure,add=T)%>%
summarise_each(funs(mean,sd,min,max),-subject) #summarise(mean=mean(score),sd=sd(score),min=min(score),max=max(score))でも同じ。もともとsummarise_eachは複数列に同じ関数を適用するときに威力を発揮するものだけど
## Source: local data frame [9 x 6]
## Groups: test [?]
##
## test measure mean sd min max
## (chr) (chr) (dbl) (dbl) (dbl) (dbl)
## 1 delayed accuracy 8.050833 1.0231177 5.5291358 10.250133
## 2 delayed complexity 6.043974 0.8633653 4.1740878 7.908483
## 3 delayed fluency 11.966818 2.2327496 6.7231359 15.657528
## 4 post accuracy 9.529745 2.4594148 2.7470626 16.876272
## 5 post complexity 7.211917 1.9710329 2.3591702 11.759346
## 6 post fluency 14.624789 3.8821630 7.3521391 21.161635
## 7 pre accuracy 6.941050 1.7114477 3.3338811 10.798454
## 8 pre complexity 4.326063 2.1271468 -0.7051241 10.391882
## 9 pre fluency 10.239550 2.0043330 5.8044643 14.556087
※このデータの場合,test,measure,score,subjectの4つしかなく,グルーピング変数で2つ使っていて,-subjectするので必然的にscoreだけの計算になるけど,もっと列数多くて記述統計出したいのは1つだけの時は列を指定してあげる(下の例参照)
追記
- もしも人ごとに1試行が1行になっているようなlong型の場合は,最初に人ごとにgroup_byすればOK
#こんな感じ
#dat2%>%
# group_by(subject)%>%
# group_by(test)%>%
# group_by(measure,add=T)%>%
# summarise_each(funs(mean,sd,min,max),score)
- もし値に欠損があるときに関数が複数あるとちょっとめんどうだけど…
#こんな感じ
#dat2%>%
# group_by(subject)%>%
# group_by(test)%>%
# group_by(measure,add=T)%>%
# summarise_each(funs(mean(.,na.rm=T),sd(.,na.rm=T),min(.,na.rm=T),max(.,na.rm=T)),score)
ハッ,なに言ってるのこの人そんなのdplyrの威力をぜ~んぜんっ活かせてないんだからね!っていうのもあるかと思うのでもっと勉強したいと思いますがとりあえずtidyrからのdplyrを外国語教育研究のありそうなデータセットの形でやってみるとこんな感じになりますよというお話でした。
参考サイト
dplyrを使いこなす!基礎編
R dplyr, tidyr でのグルーピング/集約/変換処理まとめ
なにをゆう たむらゆう。
おしまい。
20160516追記
よくよく考えてみると,そもそもCAFって指標ごとに分析するからaccuracy, fluency, complexityの3列で待ってる形じゃないと分析できなくない?
そんなときはgather->separateのあとにspreadしてあげる
dat2%>%
spread(measure,score) ->dat3 #measureごとにscoreの列を作る
head(dat3)
## subject test accuracy complexity fluency
## 1 1 delayed 7.740613 7.042510 12.961946
## 2 1 post 13.070707 8.639772 8.431656
## 3 1 pre 7.480500 4.644798 11.916630
## 4 2 delayed 7.520258 6.951231 9.921947
## 5 2 post 11.462576 6.397390 13.652658
## 6 2 pre 7.090714 6.566487 10.719751
あとは上のやり方と同じ
dat3%>%
group_by(test)%>%
summarise_each(funs(mean,sd,min,max),-subject)
## Source: local data frame [3 x 13]
##
## test accuracy_mean complexity_mean fluency_mean accuracy_sd
## (chr) (dbl) (dbl) (dbl) (dbl)
## 1 delayed 8.050833 6.043974 11.96682 1.023118
## 2 post 9.529745 7.211917 14.62479 2.459415
## 3 pre 6.941050 4.326063 10.23955 1.711448
## Variables not shown: complexity_sd (dbl), fluency_sd (dbl), accuracy_min
## (dbl), complexity_min (dbl), fluency_min (dbl), accuracy_max (dbl),
## complexity_max (dbl), fluency_max (dbl)
列数が多いので表示されていないけど,出力はデータフレームなので,出力結果を何かの変数にいれてあげれば良い
ちなみに,実をいうとwide型の段階で列ごとにapplyしたほうが(ry
library(psych)
apply(dat,2,describe)
## $pre.fluency
## vars n mean sd median trimmed mad min max range skew kurtosis se
## 1 1 50 10.24 2 10.45 10.24 2.21 5.8 14.56 8.75 0 -0.6 0.28
##
## $pre.accuracy
## vars n mean sd median trimmed mad min max range skew kurtosis se
## 1 1 50 6.94 1.71 7.15 6.93 1.79 3.33 10.8 7.46 0.06 -0.43 0.24
##
## $pre.complexity
## vars n mean sd median trimmed mad min max range skew kurtosis
## 1 1 50 4.33 2.13 4.09 4.33 2.07 -0.71 10.39 11.1 0.09 0.33
## se
## 1 0.3
##
## $post.fluency
## vars n mean sd median trimmed mad min max range skew kurtosis
## 1 1 50 14.62 3.88 14.78 14.67 5.19 7.35 21.16 13.81 -0.09 -1.27
## se
## 1 0.55
##
## $post.accuracy
## vars n mean sd median trimmed mad min max range skew kurtosis
## 1 1 50 9.53 2.46 9.48 9.44 2.69 2.75 16.88 14.13 0.24 0.67
## se
## 1 0.35
##
## $post.complexity
## vars n mean sd median trimmed mad min max range skew kurtosis
## 1 1 50 7.21 1.97 6.93 7.22 2.18 2.36 11.76 9.4 -0.07 -0.31
## se
## 1 0.28
##
## $delayed.fluency
## vars n mean sd median trimmed mad min max range skew kurtosis
## 1 1 50 11.97 2.23 12.42 12.09 2.51 6.72 15.66 8.93 -0.42 -0.52
## se
## 1 0.32
##
## $delayed.accuracy
## vars n mean sd median trimmed mad min max range skew kurtosis
## 1 1 50 8.05 1.02 8 8.06 1.06 5.53 10.25 4.72 -0.15 -0.2
## se
## 1 0.14
##
## $delayed.complexity
## vars n mean sd median trimmed mad min max range skew kurtosis se
## 1 1 50 6.04 0.86 5.97 6.05 1.01 4.17 7.91 3.73 0.02 -0.65 0.12
##
## $subject
## vars n mean sd median trimmed mad min max range skew kurtosis se
## 1 1 50 25.5 14.58 25.5 25.5 18.53 1 50 49 0 -1.27 2.06